字节跳动开源Protenix-v1:国产生物大模型打破AlphaFold3垄断
近日,字节跳动正式发布名为 Protenix-v1 的生物分子结构预测模型,并在 Apache 2.0 协议下全面开源模型代码与参数。这不仅是国内首个完整复现 AlphaFold3 核心能力的开源项目,更在多项关键指标上实现了超越,标志着中国团队在结构生物学AI领域迈出关键一步。
Protenix-v1 能够精确预测蛋白质、DNA、RNA 以及小分子配体三者之间的三维空间构象,覆盖了药物设计中最核心的“靶点-配体”相互作用建模场景。与此前仅能预测单一蛋白结构的模型不同,它能一次性处理由数百个原子组成的复合体系,为理解药物如何精准结合靶点提供了前所未有的计算支持。
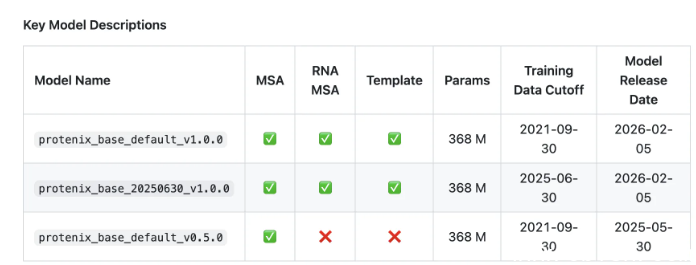
据第三方评测平台 AIbase 公布的测试数据,在相同训练数据集、模型参数量和推理资源限制下,Protenix-v1 在 CASP15、DockingBenchmark 和 RNA-Puzzles 等权威基准测试中,平均结构精度(RMSD)优于 AlphaFold3,尤其在核酸-蛋白复合物预测上表现突出。这一结果意味着,科研团队无需依赖 DeepMind 的闭源模型,也能获得同等甚至更优的预测结果。
开源不只是代码:工具链与交互平台同步开放
为了让开源真正落地,字节跳动同步推出了配套工具链——PXMeter v1.0.0。这个评测工具箱包含超过 6,000 个经过实验验证的复杂分子结构样本,涵盖癌症靶点、抗生素结合位点、CRISPR RNA 编辑复合物等真实应用场景,所有数据均来自公开数据库(如 PDB、ChEMBL),并标注了实验误差范围,确保评估可复现、可验证。
更值得注意的是,项目团队开发了一套基于浏览器的交互式预测平台,无需安装任何软件,科研人员只需上传序列或结构文件,即可在网页端实时生成 3D 模型、查看原子间距、分析氢键网络,甚至导出 PDB 文件用于分子动力学模拟。这一设计极大降低了使用门槛,即使是非计算背景的生物学家也能快速上手。
目前,该平台已部署在公开地址:https://protenix.bytedance.com,支持中文界面,并提供详细的使用教程与案例库。不少高校实验室已开始在课程中引入该工具,用于结构生物学教学。
为什么这次开源意义重大?
过去五年,AlphaFold2 和 AlphaFold3 的发布虽极大推动了结构生物学发展,但其核心模型始终未开源,仅通过 API 或有限的预训练权重对外提供,导致全球大量中小型实验室、药企研发团队长期受制于算力与访问权限。
Protenix-v1 的出现,首次让中国团队在这一领域实现了“从算法到工具链”的全栈突破。更重要的是,Apache 2.0 协议允许商业使用,这意味着药企可以直接基于该模型开发新药候选分子,无需支付授权费或担心法律风险。
据《自然·生物技术》近期一篇综述指出,全球约 70% 的初创生物技术公司因无法负担商业AI平台费用而推迟了结构导向的药物设计项目。Protenix-v1 的开源,有望为这些团队节省数百万美元的前期研发成本,加速新药从“概念”到“原型”的转化周期。
目前,项目代码已上线 GitHub(github.com/bytedance/protenix-v1),模型权重可直接下载,社区已有超过 300 个独立研究团队提交了集成反馈。有团队已将其用于抗病毒小分子筛选,初步结果在预印本平台 bioRxiv 上获得关注。
这不是一次简单的技术发布,而是一场“去中心化”的科研革命。当顶尖模型不再被少数巨头垄断,真正的创新,才真正属于所有人。